DP4ML - Feature Selection - Phần 2 - Select Categorical Input Features
Bài thứ 9 trong chuỗi các bài viết về chủ đề Data Preparation cho các mô hình ML và là bài thứ 2 về Feature Selection. Trong bài này, chúng ta sẽ tìm hiểu phương pháp lựa chọn features đối với dữ liệu có Input Variance thuộc kiểu Categorical thông qua việc thực hành trên một bộ dữ liệu cụ thể.
1. Breast Cancer Categorical Dataset
Breast Cancer Categorical Dataset là bộ dữ liệu thường được dùng cho việc nghiên cứu các thuật toán ML từ thập niên 80s. Nó bao gồm 286 mẫu dữ liệu đại diện cho các bệnh nhân, và 9 Input Variables. Thông tin về các Input Variables được miêu tả ở đây. Đây là bài toán Binary Classification, mỗi bệnh nhân thuộc 1 trong 2 nhãn là có bệnh hoặc không có bệnh.
Code dưới đây đọc vào dữ liệu, in ra một vài samples, chia dữ liệu thành Input/Output Variables, sau đó lại chia thành 2 tập train/test.
# load the dataset
def load_dataset(filename):
# load the dataset
data = read_csv(filename, header=None)
# display some samples
print(data.head())
# retrieve array
dataset = data.values
# split into input and output variables
X = dataset[:, :-1]
y = dataset[:,-1]
# format all fields as string
X = X.astype(str)
return X, y
# load the dataset
X, y = load_dataset( ' breast-cancer.csv ' )
# split into train and test sets
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.33, random_state=1)
# summarize
print( ' Train ' , X_train.shape, y_train.shape
print( ' Test ' , X_test.shape, y_test.shape)
Kết quả thực hiện:
0 1 2 3 4 5 6 7 8 9
0 '40-49' 'premeno' '15-19' '0-2' 'yes' '3' 'right' 'left_up' 'no' 'recurrence-events'
1 '50-59' 'ge40' '15-19' '0-2' 'no' '1' 'right' 'central' 'no' 'no-recurrence-events'
2 '50-59' 'ge40' '35-39' '0-2' 'no' '2' 'left' 'left_low' 'no' 'recurrence-events'
3 '40-49' 'premeno' '35-39' '0-2' 'yes' '3' 'right' 'left_low' 'yes' 'no-recurrence-events'
4 '40-49' 'premeno' '30-34' '3-5' 'yes' '2' 'left' 'right_up' 'no' 'recurrence-events'
Train (191, 9) (191,)
Test (95, 9) (95,)
2. Transform Data
Có thể nhận ra rằng, tất cả Input Variables đều có dạng Categorical. Vì thế chúng ta cần chuyển chúng sang dạng Integer để model có thể học được. Lớp OridinalEncoder trong Scikit-learn giúp chúng ta thực hiện việc này.
# prepare input data
def prepare_inputs(X_train, X_test):
oe = OrdinalEncoder()
oe.fit(X_train)
X_train_enc = oe.transform(X_train)
X_test_enc = oe.transform(X_test)
return X_train_enc, X_test_enc
Output Variable cũng phải chuyển sang Integer. Vì đây là bài toán Binary Classification nên ánh xạ hai nhãn thành 0 và 1. Sử dụng lớp LabelEncoder của Scikit-learn để thực hiện.
# prepare target
def prepare_targets(y_train, y_test):
le = LabelEncoder()
le.fit(y_train)
y_train_enc = le.transform(y_train)
y_test_enc = le.transform(y_test)
return y_train_enc, y_test_enc
Code đầy đủ như sau:
# example of loading and preparing the breast cancer dataset
from pandas import read_csv
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import LabelEncoder
from sklearn.preprocessing import OrdinalEncoder
# load the dataset
def load_dataset(filename):
# load the dataset
data = read_csv(filename, header=None)
# retrieve array
dataset = data.values
# split into input and output variables
X = dataset[:, :-1]
y = dataset[:,-1]
# format all fields as string
X = X.astype(str)
return X, y
# prepare input data
def prepare_inputs(X_train, X_test):
oe = OrdinalEncoder()
oe.fit(X_train)
X_train_enc = oe.transform(X_train)
X_test_enc = oe.transform(X_test)
return X_train_enc, X_test_enc
# prepare target
def prepare_targets(y_train, y_test):
le = LabelEncoder()
le.fit(y_train)
y_train_enc = le.transform(y_train)
y_test_enc = le.transform(y_test)
return y_train_enc, y_test_enc
# load the dataset
X, y = load_dataset('breast-cancer.csv')
# split into train and test sets
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.33, random_state=1)
# prepare input data
X_train_enc, X_test_enc = prepare_inputs(X_train, X_test)
# prepare output data
y_train_enc, y_test_enc = prepare_targets(y_train, y_test)
# summarize
print('Train', X_train_enc.shape, y_train_enc.shape)
print('Test', X_test_enc.shape, y_test_enc.shape)
3. Categorical Feature Selection
Có 2 kỹ thuật có thể áp dụng cho Input Variables dạng Categorical trong việc Feature Selection là Chi-Squared Statistic và Mutual Information Statistic.
3.1 Chi-Squared Statistic
Chi-Squared Statistic - Kiểm định giả thuyết thống kê chi bình phương ($\chi^2$) là một ví dụ về kiểm định tính độc lập giữa các biến phân loại. Kết quả của thử nghiệm này có thể được sử dụng để lựa chọn các Input Variables. Nếu Input Variables độc lập với Output Variables thì chúng có thể bị xóa khỏi tập dữ liệu.
Scikit-learn thực hiện Chi-Squared Statistic thông qua hàm chi2(). Kết hợp với hàm SelectKBest(), ta có thể lựa chọn được các features mong muốn như code dưới đây:
...
fs = SelectKBest(score_func=chi2, k='all')
fs.fit(X_train, y_train)
X_train_fs = fs.transform(X_train)
X_test_fs = fs.transform(X_test)
Muốn biêt và so sánh Score của mỗi feature, chúng ta có thể thể hiện chúng lên đồ thị. Score càng lớn càng tốt.
...
# what are scores for the features
for i in range(len(fs.scores_)):
print('Feature %d: %f' % (i, fs.scores_[i]))
# plot the scores
pyplot.bar([i for i in range(len(fs.scores_))], fs.scores_)
pyplot.show()
Code đầy đủ:
# example of chi squared feature selection for categorical data
from pandas import read_csv
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import LabelEncoder
from sklearn.preprocessing import OrdinalEncoder
from sklearn.feature_selection import SelectKBest
from sklearn.feature_selection import chi2
from matplotlib import pyplot
# load the dataset
def load_dataset(filename):
# load the dataset as a pandas DataFrame
data = read_csv(filename, header=None)
# retrieve numpy array
dataset = data.values
# split into input (X) and output (y) variables
X = dataset[:, :-1]
y = dataset[:,-1]
# format all fields as string
X = X.astype(str)
return X, y
# prepare input data
def prepare_inputs(X_train, X_test):
oe = OrdinalEncoder()
oe.fit(X_train)
X_train_enc = oe.transform(X_train)
X_test_enc = oe.transform(X_test)
return X_train_enc, X_test_enc
# prepare target
def prepare_targets(y_train, y_test):
le = LabelEncoder()
le.fit(y_train)
y_train_enc = le.transform(y_train)
y_test_enc = le.transform(y_test)
return y_train_enc, y_test_enc
# feature selection
def select_features(X_train, y_train, X_test):
fs = SelectKBest(score_func=chi2, k='all')
fs.fit(X_train, y_train)
X_train_fs = fs.transform(X_train)
X_test_fs = fs.transform(X_test)
return X_train_fs, X_test_fs, fs
# load the dataset
X, y = load_dataset('breast-cancer.csv')
# split into train and test sets
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.33, random_state=1)
# prepare input data
X_train_enc, X_test_enc = prepare_inputs(X_train, X_test)
# prepare output data
y_train_enc, y_test_enc = prepare_targets(y_train, y_test)
# feature selection
X_train_fs, X_test_fs, fs = select_features(X_train_enc, y_train_enc, X_test_enc)
# what are scores for the features
for i in range(len(fs.scores_)):
print('Feature %d: %f' % (i, fs.scores_[i]))
# plot the scores
pyplot.bar([i for i in range(len(fs.scores_))], fs.scores_)
pyplot.show()
Kết quả thực hiện:
Feature 0: 0.472553
Feature 1: 0.029193
Feature 2: 2.137658
Feature 3: 29.381059
Feature 4: 8.222601
Feature 5: 8.100183
Feature 6: 1.273822
Feature 7: 0.950682
Feature 8: 3.699989
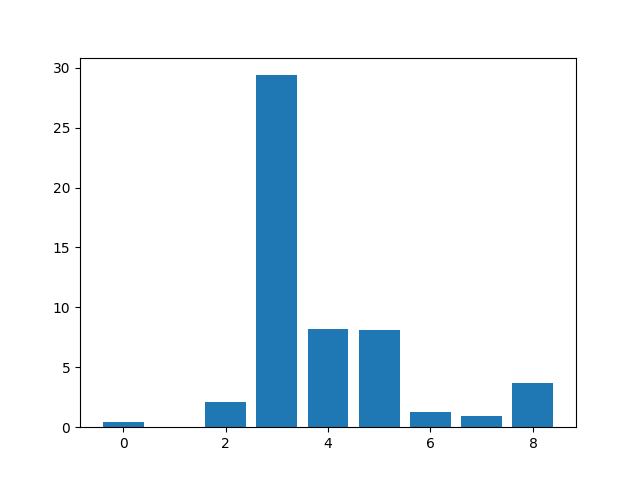
Theo kết quả này thì có lẽ 4/9 feature liên quan nhất đến Output Variables là các features 3, 4, 5, 8. Do đó, ta sẽ chọn k=4 để lấy ra 4 features này.
3.2 Mutual Information Feature Selection
Mutual Information xuất phát từ lĩnh vực Lý thuyết Thông tin. Nó đo đặc mức độ suy giảm của một biến khi đã biết giá trị của một biến khác. Trong Scikit-learn, Mutual Information được implement trong hàm mutual info classif(). Cách sử dụng cũng tương tự như Chi-Squared Statistic.
# feature selection
def select_features(X_train, y_train, X_test):
fs = SelectKBest(score_func=mutual_info_classif, k='all')
fs.fit(X_train, y_train)
X_train_fs = fs.transform(X_train)
X_test_fs = fs.transform(X_test)
return X_train_fs, X_test_fs, fs
Kết quả thực hiện:
Feature 0: 0.000000
Feature 1: 0.045906
Feature 2: 0.063114
Feature 3: 0.018165
Feature 4: 0.007357
Feature 5: 0.041046
Feature 6: 0.049755
Feature 7: 0.000000
Feature 8: 0.024087

Theo Mutual Information thì các features 2, 3, 5, 6 có mức độ liên quan đến Output Variables nhất. Chọn k=4.
4. Modeling With Selected Features
Sử dụng 2 kỹ thuật bên trên, chúng ta sẽ đi đánh giá độ chính xác của Logistic Regression model trên tập dữ liệu Breast Cancer Categorical Dataset.
4.1 Model Built using All Features
Đầu tiên, chúng ta sẽ xây dựng Base model làm cơ sở để so sánh, đánh giá. Các model về sau được mong đợi phải có độ chính xác cao hơn độ chính xác của Base model này.
# evaluation of a model using all input features
from pandas import read_csv
from sklearn.preprocessing import LabelEncoder
from sklearn.preprocessing import OrdinalEncoder
from sklearn.model_selection import train_test_split
from sklearn.linear_model import LogisticRegression
from sklearn.metrics import accuracy_score
# load the dataset
def load_dataset(filename):
# load the dataset as a pandas DataFrame
data = read_csv(filename, header=None)
# retrieve numpy array
dataset = data.values
# split into input (X) and output (y) variables
X = dataset[:, :-1]
y = dataset[:,-1]
# format all fields as string
X = X.astype(str)
return X, y
# prepare input data
def prepare_inputs(X_train, X_test):
oe = OrdinalEncoder()
oe.fit(X_train)
X_train_enc = oe.transform(X_train)
X_test_enc = oe.transform(X_test)
return X_train_enc, X_test_enc
# prepare target
def prepare_targets(y_train, y_test):
le = LabelEncoder()
le.fit(y_train)
y_train_enc = le.transform(y_train)
y_test_enc = le.transform(y_test)
return y_train_enc, y_test_enc
# load the dataset
X, y = load_dataset('breast-cancer.csv')
# split into train and test sets
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.33, random_state=1)
# prepare input data
X_train_enc, X_test_enc = prepare_inputs(X_train, X_test)
# prepare output data
y_train_enc, y_test_enc = prepare_targets(y_train, y_test)
# fit the model
model = LogisticRegression(solver='lbfgs')
model.fit(X_train_enc, y_train_enc)
# evaluate the model
yhat = model.predict(X_test_enc)
# evaluate predictions
accuracy = accuracy_score(y_test_enc, yhat)
print('Accuracy: %.2f' % (accuracy*100))
Kết quả:
Accuracy: 75.79
4.2 Model Built Using Chi-Squared Features
# evaluation of a model fit using chi squared input features
from pandas import read_csv
from sklearn.preprocessing import LabelEncoder
from sklearn.preprocessing import OrdinalEncoder
from sklearn.feature_selection import SelectKBest
from sklearn.feature_selection import chi2
from sklearn.model_selection import train_test_split
from sklearn.linear_model import LogisticRegression
from sklearn.metrics import accuracy_score
# load the dataset
def load_dataset(filename):
# load the dataset as a pandas DataFrame
data = read_csv(filename, header=None)
# retrieve numpy array
dataset = data.values
# split into input (X) and output (y) variables
X = dataset[:, :-1]
y = dataset[:,-1]
# format all fields as string
X = X.astype(str)
return X, y
# prepare input data
def prepare_inputs(X_train, X_test):
oe = OrdinalEncoder()
oe.fit(X_train)
X_train_enc = oe.transform(X_train)
X_test_enc = oe.transform(X_test)
return X_train_enc, X_test_enc
# prepare target
def prepare_targets(y_train, y_test):
le = LabelEncoder()
le.fit(y_train)
y_train_enc = le.transform(y_train)
y_test_enc = le.transform(y_test)
return y_train_enc, y_test_enc
# feature selection
def select_features(X_train, y_train, X_test):
fs = SelectKBest(score_func=chi2, k=4)
fs.fit(X_train, y_train)
X_train_fs = fs.transform(X_train)
X_test_fs = fs.transform(X_test)
return X_train_fs, X_test_fs
# load the dataset
X, y = load_dataset('breast-cancer.csv')
# split into train and test sets
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.33, random_state=1)
# prepare input data
X_train_enc, X_test_enc = prepare_inputs(X_train, X_test)
# prepare output data
y_train_enc, y_test_enc = prepare_targets(y_train, y_test)
# feature selection
X_train_fs, X_test_fs = select_features(X_train_enc, y_train_enc, X_test_enc)
# fit the model
model = LogisticRegression(solver='lbfgs')
model.fit(X_train_fs, y_train_enc)
# evaluate the model
yhat = model.predict(X_test_fs)
# evaluate predictions
accuracy = accuracy_score(y_test_enc, yhat)
print('Accuracy: %.2f' % (accuracy*100))
Kết quả:
Accuracy: 74.74
4.3 Model Built Using Mutual Information Features
# evaluation of a model fit using mutual information input features
from pandas import read_csv
from sklearn.preprocessing import LabelEncoder
from sklearn.preprocessing import OrdinalEncoder
from sklearn.feature_selection import SelectKBest
from sklearn.feature_selection import mutual_info_classif
from sklearn.model_selection import train_test_split
from sklearn.linear_model import LogisticRegression
from sklearn.metrics import accuracy_score
# load the dataset
def load_dataset(filename):
# load the dataset as a pandas DataFrame
data = read_csv(filename, header=None)
# retrieve numpy array
dataset = data.values
# split into input (X) and output (y) variables
X = dataset[:, :-1]
y = dataset[:,-1]
# format all fields as string
X = X.astype(str)
return X, y
# prepare input data
def prepare_inputs(X_train, X_test):
oe = OrdinalEncoder()
oe.fit(X_train)
X_train_enc = oe.transform(X_train)
X_test_enc = oe.transform(X_test)
return X_train_enc, X_test_enc
# prepare target
def prepare_targets(y_train, y_test):
le = LabelEncoder()
le.fit(y_train)
y_train_enc = le.transform(y_train)
y_test_enc = le.transform(y_test)
return y_train_enc, y_test_enc
# feature selection
def select_features(X_train, y_train, X_test):
fs = SelectKBest(score_func=mutual_info_classif, k=4)
fs.fit(X_train, y_train)
X_train_fs = fs.transform(X_train)
X_test_fs = fs.transform(X_test)
return X_train_fs, X_test_fs
# load the dataset
X, y = load_dataset('breast-cancer.csv')
# split into train and test sets
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.33, random_state=1)
# prepare input data
X_train_enc, X_test_enc = prepare_inputs(X_train, X_test)
# prepare output data
y_train_enc, y_test_enc = prepare_targets(y_train, y_test)
# feature selection
X_train_fs, X_test_fs = select_features(X_train_enc, y_train_enc, X_test_enc)
# fit the model
model = LogisticRegression(solver='lbfgs')
model.fit(X_train_fs, y_train_enc)
# evaluate the model
yhat = model.predict(X_test_fs)
# evaluate predictions
accuracy = accuracy_score(y_test_enc, yhat)
print('Accuracy: %.2f' % (accuracy*100))
Kết quả:
Accuracy: 76.84
Theo các kết quả thu được từ 3 lần thí nghiệm thì có thể thấy rằng Mutual Information mang lại kết quả tốt nhất. Để kết quả được tin cậy hơn, bạn có thể sử dụng k-Fold Cross-Validation thay vì chỉ chia Train/Test thông thường.
5. Kết luận
Bài thứ 2 về chủ đề Feature Selection, mình đã giới thiệu 2 kỹ thuật Chi-Squared Statistic và Mutual Information áp dụng cho Input Variables dạng Categorical.
Toàn bộ code của bài này, các bạn có thể tham khảo tại đây.
Bài tiếp theo sẽ là các kỹ thuật Feature Selection áp dụng cho Input Variables dạng Numerical. Mời các bạn đón đọc.
6. Tham khảo
[1] Jason Brownlee, “Data Preparation for Machine Learning”, Book: https://machinelearningmastery.com/data-preparation-for-machine-learning/.